Microbiología Estructural y Biodiseño

Resumen
Estudiamos la estructura tridimensional de componentes esenciales de la materia viva, a fin de arribar a una comprensión a resolución atómica de la arquitectura y los mecanismos moleculares de la vida. Nos enfocamos en la investigación de las bases moleculares de la actividad de proteínas relevantes para el fitness bacteriano, en particular en sistemas de transducción de señales en bacterias patógenas. Estos sistemas evolucionaron para mediar la adaptación de la fisiología bacteriana al entorno en el hospedador y la sobrevida frente a la reacción del sistema inmune. Entones, nos proponemos comprender de qué manera las interfaces proteína:proteína (co)evolucionan para mediar el flujo de información en bacterias y servir como fuerza motriz en la evolución de la vida. Empleamos enfoques interdisciplinarios, integrando biología estructural, bioquímica y biofísica de proteínas, junto con abordajes computacionales y ensayos en in vivo.
Líneas de Investigación
Mecanismos moleculares de la regulación del metabolismo de nitrógeno en Actinobacteria
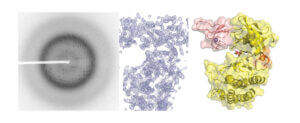
Hemos contribuido a demostrar que en diversas actinobacterias el metabolismo de nitrógeno es regulado por la vía de señalización que involucra a la quinasa de proteínas PknG. Tanto en el patógeno que causa tuberculosis en humanos como en especies de vida libre empleadas en procesos biotecnológicos, PknG controla los niveles intracelulares de glutamato en función de los aminoácidos presentes en el medio. Notablemente, la integridad de esta vía de transducción de señales es crítica para la virulencia del bacilo de Koch. En Mycobacterium el regulador GarA modula por interacción directa tres enzimas metabólicas, entre ellas una glutamato deshidrogenasa de alto peso molecular (L-GDH, L por large, en inglés). Esto determina que el alfa-cetoglutarato se desvíe del ciclo de Krebs en favor de la síntesis de glutamato; en cambio, la fosforilación de GarA por PknG inhibe la acción del regulador y el alfa-cetoglutarato es entonces direccionado al ciclo de Krebs. Recientemente elucidamos la estructura 3D de la L-GDH micobacteriana modulada por GarA. Estos hallazgos revelaron aspectos únicos de la arquitectura de este tipo de enzimas y sugieren mecanismos de regulación novedosos. Una comprensión profunda de los mecanismos moleculares involucrados en esta vía de señalización permitirá descifrar cómo M. tuberculosis y otras actinobacterias modulan su metabolismo y demás procesos fisiológicos claves. Esto aportará al diseño de drogas contra la tuberculosis y de innovaciones biotecnológicas.
SERVICIOS
Se brindan servicios tecnológicos a través de la Plataforma Argentina de Biología Estructural y Metabolómica PLABEM.
Publicaciones Seleccionadas
- 3D architecture and structural flexibility revealed in the subfamily of large glutamate dehydrogenases by a mycobacterial enzyme. Communications Biology 4, 684. Lázaro, M., Melero, R., Huet, C., López-Alonso, J.P., Delgado, S., Dodu, A., Bruch, E.M., Abriata, L.A., Alzari, P.M., Valle, M. and Lisa, M.N. (2021). https://doi.org/10.1038/s42003-021-02222-x
- A Tetratricopeptide Repeat Scaffold Couples Signal Detection to OdhI Phosphorylation in Metabolic Control by the Protein Kinase PknG. mBio 12, e0171721. Lisa, M.N., Sogues, A., Barilone, N., Baumgart, M., Gil, M., Graña, M., Durán, R., Biondi, R., Bellinzoni, M., Bott, M., Alzari, P.M. (2021). https://doi.org/10.1128/mBio.01717-21
- PknG senses amino acid availability to control metabolism and virulence of Mycobacterium tuberculosis. PLoS Pathogens 13, e1006399. Rieck, B., Degiacomi, G., Zimmermann, M., Cascioferro, A., Boldrin, F., Lazar-Adler, N.R., Bottrill, A.R., le Chevalier, F., Frigui, W., Bellinzoni, M., Lisa, M.N., Alzari, P.M., Nguyen, L., Brosch, R., Sauer, U., Manganelli, R. and O’Hare, H. (2017). https://doi.org/10.1371/journal.ppat.1006399
- Molecular Basis of the Activity and the Regulation of the Eukaryotic-like S/T Protein Kinase PknG from Mycobacterium tuberculosis. Structure 23, 1039-1048. Lisa, M.N., Gil, M., André-Leroux, G., Barilone, N., Durán, R., Biondi, R.M. and Alzari, P.M. (2015). https://doi.org/10.1016/j.str.2015.04.001
For a complete list, check my orcid.org/0000-0001-9630-8090
TW: @MaNataliaLisa