GRUPOS DE INVESTIGACIÓN
Estructura, Plegamiento y Función de Proteínas
RESUMEN

Sede:
Email:
- Berrocal, Rodrigo
- Bertero, Fabricio
- Bianchimano Macat, Luciana
- Don, Carla
- Garavaglia, Macarena
LÍNEA DE INVESTIGACIÓN
Estudio de las relaciones estructura-función de chaperones moleculares
El proteoma celular resulta del balance entre la síntesis de proteínas y su degradación. El destino de la célula depende de la capacidad para mantener la homeostasis proteica (proteostasis). Un desequilibrio del proteoma apropiado para una situación puede producir daños importantes a la célula, incluso su muerte.
Múltiples mecanismos responden a las variaciones del proteoma mediante sistemas regulados. En conjunto, estos mecanismos se conocen como “control de calidad de proteínas” y consisten en un entramado de chaperones moleculares, proteasas y proteínas accesorias que eliminan proteínas dañadas, solubilizan agregados proteicos potencialmente tóxicos o asisten en el plegamiento de polipéptidos.
En ciertas situaciones, el control de calidad de proteínas puede verse sobrecargado. Por eso, en los distintos compartimentos celulares se dispara un programa genético en donde la expresión de los miembros de la red aumenta para contrarrestar el estrés. Este proceso se conoce como respuesta a proteínas desplegadas o UPR (unfolded protein response).
Nuestro grupo estudia las condiciones que gatillan la UPR, como se comunica el estrés al núcleo y que procesos de defensa moleculares se activan en la célula para asegurar su sobrevida. Utilizamos técnicas como proteómica, western blot, transformación vegetal y resonancia magnética nuclear, entre otras. Responsables: Dr. Germán Rosano / Dr. Eduardo Ceccarelli.
Estudio de las relaciones estructura-función de flavoenzimas
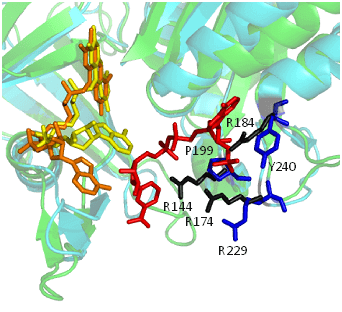
Las ferredoxina-NADP+ reductasas (FNR) son proteínas monoméricas, que contienen FAD unido no covalentemente como grupo prostético. Estas flavoenzimas, ampliamente distribuidas, intervienen en la transferencia de electrones de múltiples procesos biológicos importantes. Las FNR catalizan la transferencia reversible de electrones entre el NADP(H) y transportadores mono-electrónicos (ferredoxina o flavodoxina). Se clasifican en FNR tipo planta o mitocondrial. Las FNR tipo planta se dividen a su vez en plastíticas y bacterianas. Estas últimas participan en vías metabólicas apropiadas para el desarrollo de antimicrobianos por no estar presentes en humanos. Así, pueden emplearse para diseñar inhibidores destinados a combatir enfermedades causadas por diferentes patógenos.
Nuestros estudios se dirigen a comprender el mecanismo catalítico de las FNR de plantas y bacterias, los procesos metabólicos relevantes en los que participan y las características estructurales que definen la eficiencia catalítica de estas enzimas. Nos abocamos al análisis estructural y funcional de las FNR y la identificación y caracterización de sus sustratos naturales.
El acercamiento experimental incluye el análisis de enzimas recombinantes salvajes y modificadas por ingeniería de proteínas. Empleamos análisis cinéticos, métodos biofísicos y estructurales, y de modelización. Responsables: Dra. Daniela Catalano Dupuy / Dr. Eduardo Ceccarelli.
IMÁGENES DE NUESTRAS INVESTIGACIONES


PUBLICACIONES Y PATENTES
Structural features of the plant N-recognin ClpS1 and sequence determinants in its targets that govern substrate selection.
Structural features of the plant N-recognin ClpS1 and sequence determinants in its targets that govern substrate selection.
A new catalytic mechanism of bacterial ferredoxin‐NADP+ reductases due to a particular NADP+ binding mode.
A new catalytic mechanism of bacterial ferredoxin‐NADP+ reductases due to a particular NADP+ binding mode.
Biochemical characterization of ClpB3, a chloroplastic disaggregase from Arabidopsis thaliana.
Biochemical characterization of ClpB3, a chloroplastic disaggregase from Arabidopsis thaliana.
A bacterial [4Fe-4S] ferredoxin as redox partner of the plastidic-type ferredoxin-NADP+ reductase from Leptospira interrogans.
A bacterial [4Fe-4S] ferredoxin as redox partner of the plastidic-type ferredoxin-NADP+ reductase from Leptospira interrogans.
New tools for recombinant protein production in Escherichia coli: a 5-year update.
New tools for recombinant protein production in Escherichia coli: a 5-year update.
Sede CCT Rosario
Ocampo y Esmeralda, Predio CONICET-Rosario
2000 Rosario, Santa Fe, Argentina
Tel. 54-341-4237070 / 4237500 / 4237200
Sede Facultad de Ciencias Bioquímicas y Farmacéuticas
Universidad Nacional de Rosario - Suipacha 531
2000 Rosario, Santa Fe, Argentina
Tel. +54 341 4350596 / 4350661 / 4351235
#diadelamujerylaninaenlaciencia
Celebramos este día en la voz de nuestra investigadora de ingreso mas reciente en IBR!



