GRUPOS DE INVESTIGACIÓN
Sensores Bacterianos
RESUMEN

LÍNEA DE INVESTIGACIÓN
Mecanismo de transducción de señal de las proteínas BlaR1 y MecR1 de Staphylococcus aureus
Staphylococcus aureus es la principal causa de infecciones intra- y extra-hospitalarias en el mundo. Existe gran preocupación por la creciente prevalencia de infecciones causadas por cepas de S. aureus resistentes a la acción de los antibióticos β-lactámicos (MRSA, S. aureus meticilino-resistente). Los mecanismos de resistencia a los antibióticos β-lactámicos en S. aureus se activan solo cuando la bacteria se encuentra en presencia de un β-lactámico. Esta activación está regulada por las proteínas transmembrana sensoras/transductoras de señales BlaR1 y MecR1. MecR1 regula la expresión de la transpeptidasa PBP2a, no inhibida por concentraciones clínicas de la mayoría de estos antibióticos. En tanto que BlaR1 regula la expresión de la serin-β-lactamasa PC1 y, en algunas cepas, de PBP2a. Nuestro grupo está abocado a caracterizar estructuralmente a MecR1 y BlaR1, para comprender los cambios conformacionales que llevan a la activación de estas metaloproteasas por β-lactámicos. Para esto combinamos ensayos microbiológicos (cepas reporteras), bioquímicos (ensayos de actividad, entrecruzamiento químico con compuestos fotoactivables, entre otros) y biofísicos (crio-microscopía electrónica, dicroísmo circular, espectroscopía de Resonancia Paramagnética Electrónica (en colaboración con el Dr. Tabares, I2BC, Francia) y Transferencia de Energía por Resonancia de Luminiscencia, entre otras técnicas).
Detalles moleculares del reconocimiento de antibióticos β-lactámicos y glicopéptidos por las proteínas sensoras del sistema VraSRT de Staphylococcus aureus
El glicopéptido vancomicina es el antibiótico disponible para el tratamiento de infecciones por MRSA. El panorama no es sin embargo alentador, dado que algunas cepas de S. aureus también han desarrollado mecanismos de resistencia a vancomicina. El sistema de tres componentes VraSRT, coordina la respuesta a antibióticos que inhiben la biosíntesis del peptidoglicano de la pared celular en S. aureus y está asociado a la resistencia a antibióticos glicopéptidos y β-lactámicos. La inactivación de este sistema afecta drásticamente la resistencia de diferentes cepas de S. aureus a vancomicina y también a antibióticos β-lactámicos. Se desconoce la señal que dispara la activación de la proteína sensora/quinasa VraS y el rol de la proteína de membrana VraT. Empleando fotosondas derivadas de antibióticos, cepas reporteras y ensayos de actividad quinasa/fosfotransferasa, estamos evaluando si la activación se debe a la interacción directa con los antibióticos. Además, abordamos la caracterización estructural de los cambios conformacionales que dan lugar a la activación, empleando crio-microscopía electrónica, dicroísmo circular, espectroscopía de Resonancia Paramagnética Electrónica (en colaboración con el Dr. Tabares, I2BC, Francia) y Transferencia de Energía por Resonancia de Luminiscencia, entre otras técnicas.
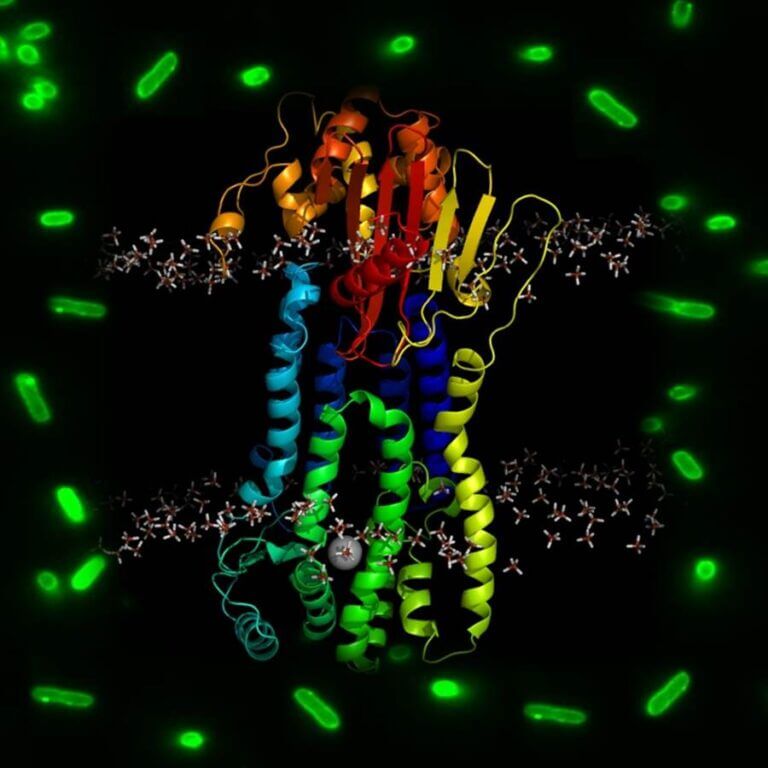
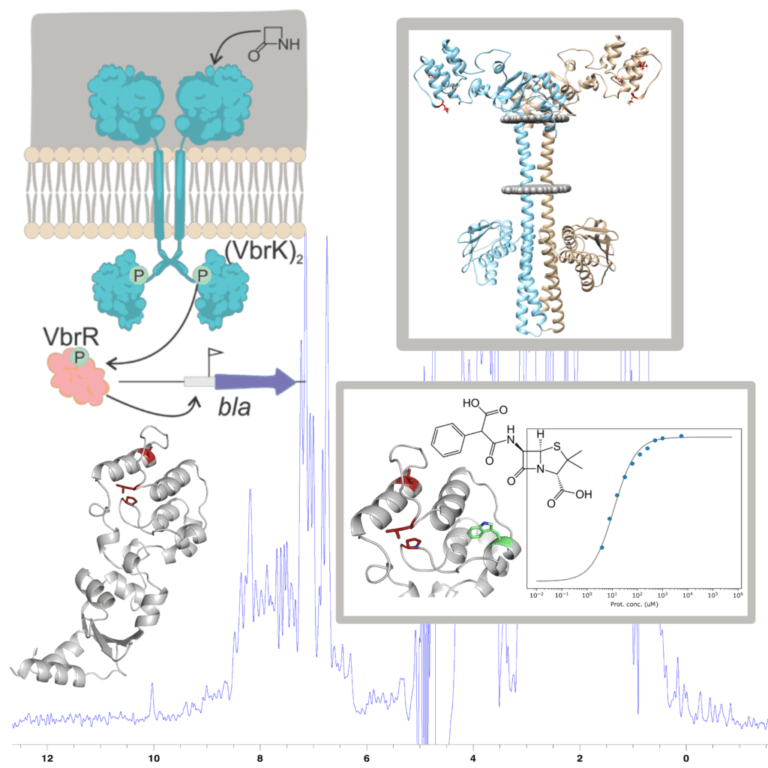
Reconocimiento de antibióticos β-lactámicos por el sistema de resistencia VbrKR de Vibrio parahaemolyticus
El surgimiento de bacterias resistentes a antibióticos β-lactámicos es un problema de salud pública a nivel mundial. Cepas resistentes del género Vibrio (causantes de infecciones intestinales agudas) han sido reportadas alrededor de todo el planeta, constituyendo una amenaza especialmente en países en desarrollo. Recientemente se ha descripto un nuevo sistema sensor directo de antibióticos β-lactámicos que activa la producción de β-lactamasa en Vibrio parahaemolyticus. Se trata de un sistema de dos componentes denominado VbrKR. Este sistema se encuentra principalmente en bacterias del género Vibrio, convirtiéndolo en un blanco específico para el tratamiento de infecciones causadas por especies de este género. Nuestro objetivo es la comprensión del mecanismo molecular de activación de VbrKR. Empleando técnicas biofísicas apuntamos a caracterizar la afinidad del receptor por distintos antibióticos, determinar su estructura por cristalografía de rayos X en presencia y ausencia del antibiótico, determinar su estado oligomérico, y generar un sistema de mapeo de interacciones por RMN. Esta información proveerá las bases para el diseño racional de drogas para tratar estas infecciones.
Identificación de inhibidores de los sensores de antibióticos β-lactámicos en Staphylococcus aureus
La resistencia a antibióticos β-lactámicos y glicopéptidos en S. aureus se debe a la expresión inducible de diferentes determinantes de resistencia. S. aureus cuenta con proteínas de membrana que son sensores de estos antibióticos. Las proteínas sensoras pueden ser metaloproteasas (como es el caso de las proteínas BlaR1 y MecR1) o quinasas de sistemas de dos/tres componentes (como es el caso de la proteína VraS). La interacción con el antibiótico desata un mecanismo de transducción de señal que lleva a la activación del dominio citoplasmático de estas proteínas (dominio catalítico), dando lugar a una cascada de reacciones en el citoplasma que culmina en la expresión de los determinantes de resistencia. En colaboración con el Dr. Rodriguez (IBR) y el Dr. Testero (IQUIR) trabajamos para identificar compuestos capaces de inhibir la activación de estos sistemas de resistencia y caracterizar su mecanismo de inhibición. El hallazgo de compuestos inhibidores proveería de información clave para comprender el mecanismo de transducción de señal. Además, estos inhibidores permitirían el diseño de terapias combinadas con β-lactámicos/glicopéptidos para el tratamiento de infecciones con cepas resistentes.
IMÁGENES DE NUESTRAS INVESTIGACIONES