GRUPOS DE INVESTIGACIÓN
Estructura y Fisiología de Biofilms Microbianos
RESUMEN

- Serra, Diego Sede: CCT
Email: dserra@ibr-conicet.gov.ar
- Obando, Mayra
- Valentinis Rossi, Franco
- Abitante, Alma
- Olazaran Mainetti, Josefina
LÍNEA DE INVESTIGACIÓN
Patrones espaciales y mecanismos de tolerancia a los antibióticos en biofilms bacterianos
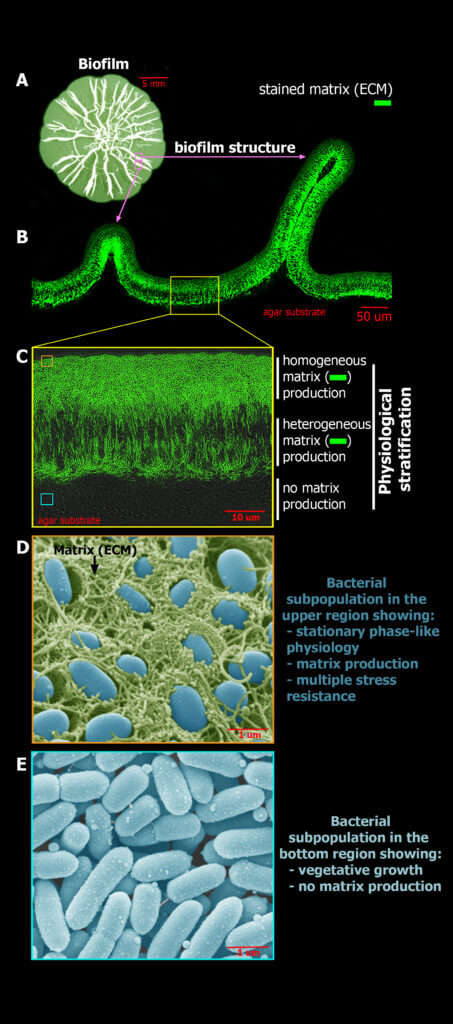
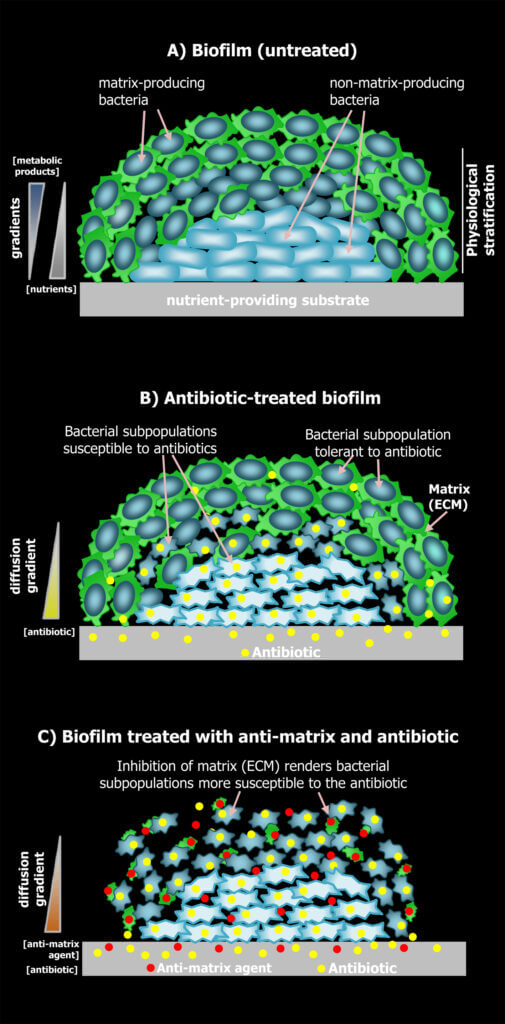
Debido a su complejidad estructural, los biofilms exhiben microambientes internos muy heterogéneos definidos por gradientes de nutrientes y productos metabólicos. Allí, las bacterias adaptan su fisiología a las condiciones de su entorno, lo que conlleva a la estratificación de los biofilms en regiones con características fisiológicas muy distintas. Nuestros trabajos sugieren que esta estratificación fisiológica no sólo representaría una división de tareas dónde subpoblaciones celulares se especializan localmente en la realización
de tareas específicas, como, por ejemplo, la producción de matriz, sino que también dotaría a las subpoblaciones de diferentes capacidades para afrontar situaciones de estrés, como los tratamientos antibióticos. Nuestro grupo está interesado en caracterizar esta estratificación fisiológica en biofilms de cepas comensales y patógenas de E. coli y en comprender cómo esta heterogeneidad fisiológica influye en las posibilidades de sobrevida de las bacterias a los antibióticos en función de su ubicación dentro de los biofilms. En particular, nos proponemos esclarecer cómo la heterogeneidad en la producción de componentes de matriz (amiloide curli y fEtN-celulosa), en las respuestas celulares al estrés y en el metabolismo entre subpoblaciones bacterianas influye en la tolerancia antibiótica en biofilms de E. coli. El conocimiento de estos aspectos es crucial para el diseño de terapias que puedan eliminar a todas las subpoblaciones celulares que coexisten en estas comunidades, independientemente de su estado fisiológico y localización espacial.
Identificación y mecanismos de acción de compuestos anti-biofilms
Atentos a la necesidad de alternativas terapéuticas para procesos infecciosos que involucran la formación de biofilms, estamos también interesados en descubrir compuestos anti-biofilms y caracterizar los mecanismos moleculares que subyacen su acción. En particular, buscamos compuestos que puedan interferir en la producción de las fibras amiloides de curli y de fEtN-celulosa, los componentes de matriz mayoritarios y determinantes del desarrollo estructural de los biofilms de E. coli. Como plataforma experimental para la búsqueda de inhibidores nos concentramos en explorar interacciones microbianas en biofilms. Mientras que las interacciones antagónicas entre microorganismos han sido intensamente explotadas en la búsqueda de antibióticos, es decir, compuestos capaces directamente de matar o inhibir el crecimiento bacteriano, el potencial de estas interacciones como fuentes de compuestos
capaces de modular o interferir otros comportamientos bacterianos, como es la formación de biofilms, ha permanecido prácticamente inexplorado. El uso de compuestos anti-biofilms para el tratamiento de infecciones asociadas a biofilms es crucial, ya que los mismos pueden aumentar considerablemente la eficacia de los antibióticos en terapias combinadas o facilitar la erradicación de la infección por el propio sistema inmunológico del hospedador.
IMÁGENES DE NUESTRAS INVESTIGACIONES
PUBLICACIONES Y PATENTES
Bacterial multicellularity: the biology of Escherichia coli building large-scale biofilm communities.
Bacterial multicellularity: the biology of Escherichia coli building large-scale biofilm communities.
A c-di-GMP-based switch controls local heterogeneity of extracellular matrix synthesis which is crucial for integrity and morphogenesis of Escherichia coli macrocolony biofilms.
A c-di-GMP-based switch controls local heterogeneity of extracellular matrix synthesis which is crucial for integrity and morphogenesis of Escherichia coli macrocolony biofilms.
Spatial organisation of different sigma factor activities and c-di-GMP signalling within the 3D landscape of a bacterial biofilm.
Spatial organisation of different sigma factor activities and c-di-GMP signalling within the 3D landscape of a bacterial biofilm.
Phosphoethanolamine cellulose: a naturally produced chemically modified cellulose.
Phosphoethanolamine cellulose: a naturally produced chemically modified cellulose.
The green tea polyphenol EGCG inhibits Escherichia coli biofilm formation by impairing amyloid curli fibre assembly and down-regulating the biofilm regulator CsgD via the σE-dependent sRNA RybB.
The green tea polyphenol EGCG inhibits Escherichia coli biofilm formation by impairing amyloid curli fibre assembly and down-regulating the biofilm regulator CsgD via the σE-dependent sRNA RybB.
Sede CCT Rosario
Ocampo y Esmeralda, Predio CONICET-Rosario
2000 Rosario, Santa Fe, Argentina
Tel. 54-341-4237070 / 4237500 / 4237200
Sede Facultad de Ciencias Bioquímicas y Farmacéuticas
Universidad Nacional de Rosario - Suipacha 531
2000 Rosario, Santa Fe, Argentina
Tel. +54 341 4350596 / 4350661 / 4351235
🔬 El IBR suma 9 proyectos seleccionados en Investigación Orientada 2025 de @ProduccionSF y @CienciaSantaFe.
Biotecnología, salud y sostenibilidad para fortalecer el vínculo entre ciencia, innovación y desarrollo territorial.

